adjacency_matrix#
The rows and columns are ordered according to the nodes in nodelist. If nodelist is None, then the ordering is produced by G.nodes().
dtype NumPy data-type, optional
The desired data-type for the array. If None, then the NumPy default is used.
weight string or None, optional (default=’weight’)
The edge data key used to provide each value in the matrix. If None, then each edge has weight 1.
Returns : A SciPy sparse array
Adjacency matrix representation of G.
to_numpy_array to_scipy_sparse_array to_dict_of_dicts adjacency_spectrum
For directed graphs, entry i,j corresponds to an edge from i to j.
If you want a pure Python adjacency matrix representation try networkx.convert.to_dict_of_dicts which will return a dictionary-of-dictionaries format that can be addressed as a sparse matrix.
For MultiGraph/MultiDiGraph with parallel edges the weights are summed. See to_numpy_array for other options.
The convention used for self-loop edges in graphs is to assign the diagonal matrix entry value to the edge weight attribute (or the number 1 if the edge has no weight attribute). If the alternate convention of doubling the edge weight is desired the resulting SciPy sparse array can be modified as follows:
>>> G = nx.Graph([(1, 1)]) >>> A = nx.adjacency_matrix(G) >>> print(A.todense()) [[1]] >>> A.setdiag(A.diagonal() * 2) >>> print(A.todense()) [[2]]
incidence_matrix#
The incidence matrix assigns each row to a node and each column to an edge. For a standard incidence matrix a 1 appears wherever a row’s node is incident on the column’s edge. For an oriented incidence matrix each edge is assigned an orientation (arbitrarily for undirected and aligning to direction for directed). A -1 appears for the source (tail) of an edge and 1 for the destination (head) of the edge. The elements are zero otherwise.
Parameters : G graph
nodelist list, optional (default= all nodes in G)
The rows are ordered according to the nodes in nodelist. If nodelist is None, then the ordering is produced by G.nodes().
edgelist list, optional (default= all edges in G)
The columns are ordered according to the edges in edgelist. If edgelist is None, then the ordering is produced by G.edges().
oriented: bool, optional (default=False)
If True, matrix elements are +1 or -1 for the head or tail node respectively of each edge. If False, +1 occurs at both nodes.
weight string or None, optional (default=None)
The edge data key used to provide each value in the matrix. If None, then each edge has weight 1. Edge weights, if used, should be positive so that the orientation can provide the sign.
Returns : A SciPy sparse array
The incidence matrix of G.
For MultiGraph/MultiDiGraph, the edges in edgelist should be (u,v,key) 3-tuples.
“Networks are the best discrete model for so many problems in applied mathematics” [1].
Неорентированные графы Python. Матрица инцидентности
где первая строка кол-во вершин и ребер,а остальные строки сами ребра, требуется вывести матрицу инцидентности данного графа, и матрицу смежности, реализовал через NumPy:
class mywindow(QtWidgets.QMainWindow): def __init__(self): self.Vertix = "" self.Edge = "" self.Vertixs = [] self.matrix = [] self.sums = [] super(mywindow, self).__init__() self.ui = Ui_MainWindow() self.ui.setupUi(self) self.ui.pushButton.clicked.connect(self.showDialog) self.ui.pushButton_5.clicked.connect(self.Find_adjacency) self.ui.pushButton_3.clicked.connect(self.digit) self.ui.pushButton_4.clicked.connect(self.isolated_uniformed) def showDialog(self): fname = QtWidgets.QFileDialog.getOpenFileName(self, 'Open file', '/home')[0] f = open(fname, 'r') with f: self.Vertexs = [ line.split() for line in f ] self.Vertex = self.Vertexs[0][0] self.Edge = self.Vertexs[0][1] del self.Vertexs[0] print(self.Vertex,self.Edge) print(self.Vertexs) def Find_Smegnosti(self): a = [] b = [] for i in self.Vertexs: a.append(int(i[0])) b.append(int(i[1])) print(a) print(b) self.matrix = np.zeros((int(self.Vertex)+1,int(self.Vertex)+1)) self.matrix[a,b] = 1 self.matrix[b,a] = 1 self.matrix = np.delete(self.matrix, (0), axis=0) self.matrix= np.delete(self.matrix, (0), axis=1 ) for i in range(len(self.matrix)): np.delete(self.matrix[i],0) self.ui.label_2.setText(str(self.matrix)) как имея ребра и матрицу смежности найти матрицу инцидентности?
Ответы (1 шт):
Фактически вам уже дана матрица инцидентности, только в сжатом виде.
Например, входные данные 4 1 нужно превратить в столбец из 6 элементов, где первый и четвёртый единицы, а остальные нули.
Проще всего создать нулевую матрицу и потом для каждого ребра пометить парой единици соответствующие вершины.
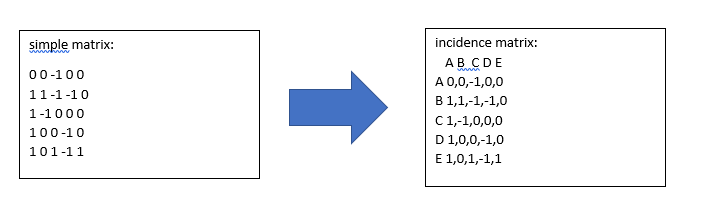
преобразовать простую матрицу в матрицу инцидентности с помощью Python
Я хочу создать графовую сеть из матрицы инцидентности, но у меня нет матрицы инцидентности, у меня есть только простая матрица. поэтому мой вопрос: как преобразовать простую матрицу в матрицу инцидентности, чтобы нарисовать сеть графов с помощью python?
2 ответа
Обе матрицы являются матрицами смежности. Самое главное знать, что это разные типы данных:
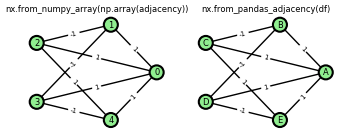
import pandas as pd import numpy as np adjacency = [[0,0,-1,0,0], [1,1,-1,-1,0], [1,-1,0,0,0], [1,0,0,-1,0], [1,0,1,-1,1]] df = pd.DataFrame(adjacency, columns = ['A','B','C','D','E'], index = ['A','B','C','D','E']) Это приводит к различным методам обработки инцидентов, а также к разной структуре графа:
Как вы легко заметите, первый метод использует автоматическое присвоение меток узлов индексам 0, 1, 2, 3, 4.
Еще один удивительный факт: вам не нужно собирать веса вручную. Они хранятся в weight атрибут кромок.
Применение:
Вы можете получить доступ к атрибутам края с помощью nx.get_edge_attributes(G, ‘weight’) . Это моя упрощенная версия структуры диаграммы:
G = nx.from_pandas_adjacency(df) pos = nx.circular_layout(G) nx.draw(G, pos, with_labels=True, bbox = dict(fc="lightgreen", ec="black", boxstyle="circle", lw=3), width=2, arrowsize=30) nx.draw_networkx_edge_labels(G, pos, edge_labels = nx.get_edge_attributes(G, 'weight')) plt.show() Надеюсь, это будет полезно, результат показан в конце
import numpy as np import networkx as nx #version 2.2 import matplotlib.pyplot as plt import pandas as pd # matrix goes here Mat = [ [0,0,-1,0,0], [1,1,-1,-1,0], [1,-1,0,0,0], [1,0,0,-1,0], [1,0,1,-1,1] ] A = pd.DataFrame(Mat) #refine rowname and colnames nodes = ["a","b","c","d","e"] A.index = nodes A.columns = nodes #create graph G0 = nx.from_pandas_adjacency(A) #create weight labels combs = <> adjc = G0.adj for i in adjc: suba = adjc[i] for j in suba: combs[(i,j)] = suba[j]['weight'] #define network structure i.e shell nx.draw_shell(G0, with_labels=True ) nx.draw_networkx_edge_labels(G0, pos=nx.shell_layout(G0), edge_labels=combs) plt.draw()